Schalter an – Schalter aus
Neue fluoreszierende Sonden verbessern Mikroskopie und medizinische Diagnostik
von Michael Schwering, Anne Seefeld und Dirk-Peter Herten
In ihrer einfachsten Ausführung werden Fluoreszenzsonden mit Licht zum Leuchten – zur Fluoreszenz – gebracht und dienen beispielsweise als Marker, um Zellstrukturen unter dem Mikroskop sichtbar zu machen. Zwischenzeitlich sind die leuchtenden Forschungswerkzeuge jedoch in raffinierter Weise weiterentwickelt worden. Die neuen Fluoreszenzsonden verbessern nicht nur die Mikroskopie und lassen das Treiben der Moleküle im Innern lebender Zellen unmittel-bar beobachten. Sie können auch die medizinische Diagnose verfeinern und beispielsweise Tumorzellen enttarnen.
Moderne fluoreszenzmikroskopische Methoden ermöglichen seit rund zwei Jahrzehnten faszinie-irende Einblicke in biologische Strukturen und Prozesse. Einzelmolekül-Studien erlauben es, die Zustände einzelner Moleküle unmittelbar zu beobachten. Damit sind nicht nur die aus Ensemblemessungen bekannten Mittelwerte zugänglich, sondern auch die Verteilung von Zuständen und deren zeitliche Abfolge. Wie sich Proteine falten, kann ebenso verfolgt werden wie die zeitliche Änderung der Aktivität einzelner Enzyme – Informationen, die bei Messungen am Molekülensemble aufgrund der Mittelung von Signalen vieler Moleküle verloren gehen. Mittlerweile entwickelte schnellere und empfindlichere Fluoreszenzmikroskope können molekulare Prozesse sogar in lebenden Zellen beobachten, beispielsweise den Transport oder die Bindung von Proteinen und Nukleinsäuren. Vor kurzem gelang es, die bis dato unüberwindbar geglaubte Grenze der optischen Auflösung von Fluoreszenzmikroskopen zu umgehen und um eine Größenordnung zu verbessern. Damit können jetzt Strukturen in der Größe einzelner Viruspartikel oder die Filamente des Zellskeletts sichtbar gemacht werden, die kleiner als 0,1 Mikrometer (tausendstel Millimeter) sind. Die genannten Errungenschaften sind hauptsächlich aus technischen Entwicklungen der Mikroskopie hervorgegangen. Inzwischen jedoch nimmt die Bedeutung von Fluoreszenzsonden immer mehr zu.
Zunächst waren Fluoreszenzsonden einfache Marker: Durch Bestrahlung mit Licht werden sie zum Leuchten, zur „Fluoreszenz“, angeregt und lassen sich dadurch spezifisch und hochempfindlich detektieren. Als Weiterentwicklung hat man Veränderungen am Molekül – sei es an dessen Struktur oder durch Wechselwirkung mit anderen Molekülen – mit Änderungen in der Emission der Fluoreszenz verknüpft. Hierzu nutzt man verschiedene photophysikalische Prozesse wie die Übertragung von Energie oder von Elektronen über kurze (atomare) Distanzen, um die Dynamik von Molekülen oder deren Bindung sichtbar zu machen.
Unsere Arbeitsgruppe entwickelt und charakterisiert Fluoreszenzsonden für den Einsatz in der Fluoreszenzmikroskopie. Wir konzentrieren uns bei unseren Untersuchungen auf dynamische Prozesse in lebenden Zellen und auf elementare chemische Reaktionsschritte, beispielsweise bei katalytischen Prozessen. Ein interessanter Nebeneffekt dieser grundlegenden Arbeiten ist die Entwicklung von Sonden, die aufgrund einer Komplexbildungsreaktion zwischen einem hellen und einem dunklen Zustand geschaltet werden können. Sie ermöglichen eine direkte Implementierung hochauflösender Mikroskopietechniken auf Basis reversibler chemischer Reaktionen.
Faszinierender Blick in die lebende Zelle
Die modernen Biowissenschaften stellen einige interessante Fragen, die möglicherweise mit geeigneten Fluoreszenzsonden in Einzelmolekülexperimenten zu beantworten sind. Vor 20 Jahren noch stand die Isolation und Charakterisierung verschiedener molekularer Zellbausteine im Vordergrund; spätestens seit der vollständigen Entschlüsselung des menschlichen Genoms im Jahr 2003 ist jedoch deutlich geworden, dass die Liste der Genbausteine alleine nicht ausreicht, um lebende Organismen und deren Reaktion auf Umwelteinflüsse zu verstehen. Vielmehr müssen die verschiedenen gleichzeitig ablaufenden Reaktionen der Komponenten – Proteine, Nukleinsäuren und Metaboliten – quantitativ erfasst werden: und zwar innerhalb der komplex strukturierten Umgebung einer lebenden Zelle.
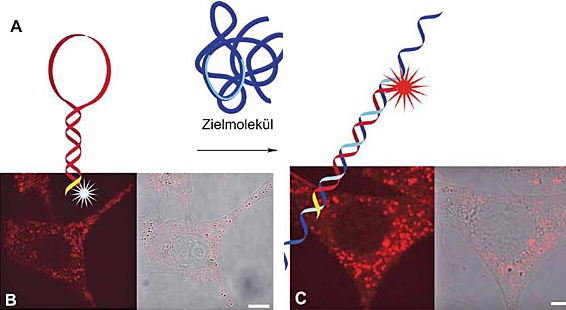
| |
|
Die Funktionsweise der Sonden: Sobald sie an ihr Zielmolekül (hier: eine Boten-Nukleinsäure) gebunden haben, beginnen sie zu leuchten (Bildreihe A oben). Die untere Bildreihe (B und C) zeigt die Ergebnisse einer Anwendung der Sonden, um Boten-Nukleinsäuren im Innern von Zellen sichtbar zu machen. In nicht stimulierten Zellen (B) tritt kaum Fluoreszenz auf, in stimulierten Zellen (C) hingegen sind mehr Zielmoleküle vorhanden, wodurch auch mehr Sonden binden können: das Fluoreszenzsignal steigt an.
|
Ein Beispiel sind Änderungen der Proteinbiosynthese als Reaktion auf äußere Einflüsse. Die Antwort der Zelle beginnt mit der Aktivierung eines Rezeptors an der Zellmembran und setzt sich über eine Signalkaskade bis in den Zellkern fort. Dort wird die Anzahl der Kopien von mRNA (Boten-Ribonukleinsäure; messenger ribonucleic acid) verändert, was in einer veränderten Anzahl von Kopien der jeweiligen Proteine resultiert.
So möchte man beispielsweise wissen, wie schnell die Zellen auf einen äußeren Stimulus reagieren oder wie lange sie brauchen, um die Produktion von mRNA zu beeinflussen. Dazu muss man messen, wie viel mRNA in Abhängigkeit von der Zeit produziert wird. Darüber hinaus ist es interessant zu erfahren, ob diese Reaktion bei allen Zellen in gleicher Weise erfolgt. Hierzu müssen die Messungen in einzelnen Zellen erfolgen.
Wir arbeiten dazu an speziellen Fluoreszenzsonden, sogenannten SmartProbes. Sie sollen es ermöglichen, mRNA in lebenden Zellen nachzuweisen. Allen Sonden ist gemeinsam, dass sie mit einem Fluoreszenzfarbstoff markiert sind. In seiner Fluoreszenz kann der Farbstoff durch Einflüsse aus seiner unmittelbaren Umgebung vermindert, also gelöscht, werden. Bei den nachfolgend beschriebenen Sonden wird die Löschung der Fluoreszenz durch einen sogenannten photoinduzierten Elektronentransfer (PET) mit der Nukleinbase Guanin erzielt.
Gensonden mit Haarnadelstruktur
Die Sonden bestehen aus einer kurzen Gensequenz und binden spezifisch über Basenpaarung an die gewünschte mRNA. Die Enden der Sonden sind selbstkomplementär: Das heißt, im freien Zustand formt die Sonde eine Haarnadelstruktur. In der Haarnadelstruktur befindet sich der Farbstoff in der Nähe von Guanin-Nukleinbasen und kann daher durch einen photoinduzierten Elektronentransfer gelöscht werden. Wenn die Sonde an die Ziel-mRNA bindet, öffnet sich die Haar-nadelstruktur – dadurch vergrößert sich der Abstand zwischen dem Farbstoff und dem zu löschenden Guanin. Die Bindung der Sonde an die mRNA wird deshalb durch eine Zunahme der Fluoreszenzintensität angezeigt.
In einem aktuellen Projekt untersuchen wir den Einfluss des Bluthormons Erythropoetin (Epo) auf das Wachstum von Lungentumoren. Lungenkrebs wird häufig erst relativ spät diagnostiziert und erfordert konservative Behandlungsmethoden wie Chemo- oder Strahlentherapie. Sowohl der Therapieerfolg als auch das Wohlbefinden der Patienten werden häufig von Blutarmut beeinträchtigt, die erheblich gebessert werden kann, wird dem Patienten blutbildendes Epo verabreicht. Die Gabe von Epo führt jedoch bei einem geringen Prozentsatz der Patienten zu einem starken Tumorwachstum. Wir untersuchen daher zurzeit in einer vom Bundesministerium für Bildung und Forschung (BMBF) geförderten Studie (LungSys) gemeinsam mit Ursula Klingmüller vom Deutschen Krebsforschungszentrum in Heidelberg und Michael Meister von der Heidelberger Thoraxklinik, ob SmartProbes eingesetzt werden können, um Patienten zu identifizieren, die eine Neigung zum Epo-abhängigen Tumorwachstum haben.
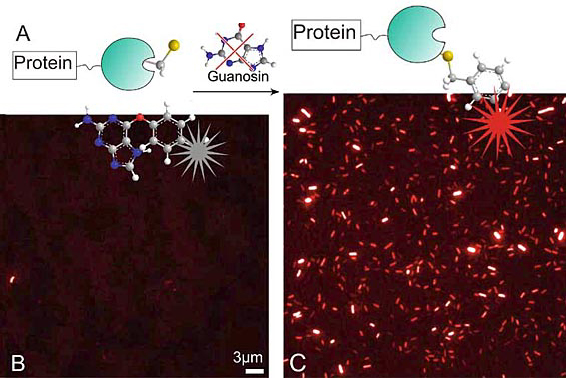
| |
|
Auch Proteine lassen sich mit fluoreszierenden Sonden markieren. Dazu wird das Erbgut einer Zelle so verändert, dass sie das Zielprotein zusammen mit einem Reparaturenzym herstellt. Das Reparaturenzym kann fluoreszente Substrate binden und mit dem Mikroskop beobachtet werden. Die mikroskopischen Bilder (B und C) zeigen Darmbakterien (Escherichia coli): Die Bakterien in Bild B enthalten das Zielprotein nicht, im Bild C hingegen ist sehr viel des Proteins enthalten.
|
Die Abbildung auf Seite 29 zeigt Zellen, die in beiden Fällen mit einer Sonde gegen eine bestimmte mRNA (Trib3) beladen wurden. Im linken Bild sind unstimulierte Zellen mit sehr niedrigem Fluoreszenzsignal – also keinen oder nur wenigen mRNA-Kopien – gezeigt. Im rechten Bild ist zu sehen, dass die Zellen eine hohe Fluoreszenzintensität aufweisen, nachdem sie mit Epo stimuliert worden sind. Die Produktion der mRNA wurde folglich in diesen Zellen durch die Gabe von Epo angeregt. Derartige Marker bergen das Potenzial, die Gen-aktivität in Tumorzellen zu messen. Sie sind damit potenzielle Kandidaten, um Tests für die medizinische Diagnostik zu entwickeln.
Leuchtende Proteine
Auch Proteine lassen sich mit Fluoreszenzsonden markieren. Dazu wird das Erbgut der Zelle so verändert, dass sie das Zielprotein zusammen mit einem Fluoreszenzprotein herstellt, welches dann mit mikroskopischen Methoden beobachtet werden kann. Ein großer Nachteil von Fluoreszenzproteinen ist, dass sie nach Bestrahlung mit Licht schnell ausbleichen, sich die Fluoreszenz dadurch also vermindert. Wir beschäftigen uns deshalb auch mit anderen Verfahren zur Markierung von Proteinen und nutzen dazu organische Farbstoffe, die exzellente photophysikalische Eigenschaften besitzen.
Es existieren bereits verschiedene, auch kommerziell erhältliche Verfahren, bei denen fluoreszierende Substrate von Zellen aufgenommen und in einer biochemischen Reaktion an bestimmte Proteine gekoppelt werden. Allerdings fluoreszieren dann sowohl das markierte Protein als auch nicht umgesetztes Substrat.
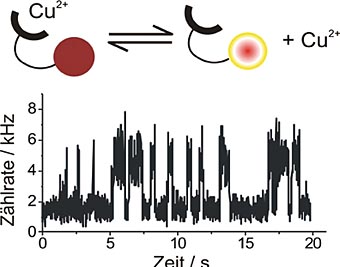
| |
|
Die Abbildung zeigt, wie die Arbeitsweise der Sonden verfeinert werden kann: Bei der Komplexierung mit einem Kupferkation wird die Fluoreszenz gelöscht. Die reversible Reaktion ist dann durch kurze und zufällige Intervalle hoher und niedriger Fluoreszenzemission gekenn-zeichnet. Eine hohe Emissionsrate entspricht dem freien Liganden, eine niedrige signalisiert den zugehörigen Kupferkomplex. Inzwischen konnten die Sonden so weiterentwickelt werden, dass sich auch Redoxreaktionen sowie katalytische Reaktionen beobachten lassen.
|
Eines dieser Verfahren basiert auf einem DNA-Reparaturenzym, das mit der Nukleinbase Guanin arbeitet. Dieser sogenannte SNAPtag überträgt fluoreszenzmarkierte Seitengruppen von der Nukleinbase Guanin auf sich selbst. Durch diese Reaktion wird in lebenden Zellen ein Fluoreszenzfarbstoff durch chemische Bindung an das Protein gebunden. Allerdings verbleiben auch hier nicht umgesetzte Guaninsubstrate in der Zelle und tragen zur Gesamtfluoreszenz bei. Wir haben deshalb verschiedene Modifikationen dieses Systems untersucht, bei denen die Fluoreszenz des Farbstoffmoleküls durch die Nukleinbase Guanin gelöscht wird. Das Ziel dabei ist, unspezifische Signale zu unterdrücken und aufwändige Waschschritte bei der Präparation der Proben zu vermeiden.
Der Vorteil dieser Markierungsmethode für Proteine ist, dass organische Farbstoffe verwendet werden können, deren Fluoreszenz „angeschaltet“ wird, sobald sie an das Protein koppeln. Die Abbildung auf Seite 30 zeigt Fluoreszenzaufnahmen des Darmbakteriums Escherichia coli (E.coli), die in beiden Fällen mit einem markierten Guaninsubstrat beladen wurden. Die E.coli im linken Bild produzieren kein Protein zur Markierung und zeigen keine unspezifische Fluoreszenz, weil das Guaninsubstrat in seiner Fluoreszenz gelöscht wird. Im rechten Bild wurden E.coli verwendet, in denen das Protein zur Markierung produziert wird. Die an das Protein gekoppelten Farbstoffmoleküle zeigen jetzt hohe, spezifische Fluoreszenzsignale. Derzeit nutzen wir diese Markierung in Zusammenarbeit mit Victor Sourjik vom Zentrum für Molekulare Biologie der Universität Heidelberg (ZMBH), um Signalkaskaden zu untersuchen, die bei der Chemotaxis der Bakterien eine wichtige Rolle spielen.
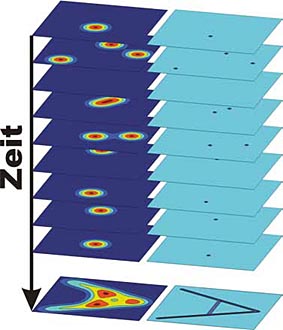
| |
|
Herkömmliche Mikroskope können Moleküle, die nahe beieinander liegen, nicht auflösen. Die Hochauflösungsmikroskopie kann dieses Problem umgehen, indem die Fluoreszenz der Moleküle ein- oder ausgeschaltet wird. Damit lässt sich die Auflösung um den Faktor 10 verbessern.
|
Elementare Schritte chemischer Reaktionen
Gemeinsam mit verschiedenen Arbeitsgruppen (R. Krämer, Anorganische Chemie, und Oliver Trapp, Organische Chemie, Heidelberg, sowie G. Jung, Biophysikalische Chemie, Universität des Saarlands) setzen wir Fluoreszenzsonden bei einer Reihe anderer Experimente ein, um elementare Reaktionsschritte chemischer Reaktionen in Einzelmolekül-Experimenten zu beobachten. Diese Arbeiten haben wir vor ein paar Jahren mit der Untersuchung der reversiblen Komplexierung von Kupfer(II)-Ionen begonnen. Hier konnten wir erstmals eine reversible chemische Reaktion direkt an einzelnen Molekülen beobachten.
Die Abbildung auf Seite 31 oben zeigt das Schema der Reaktion, bei der die Fluoreszenz bei Komplexierung eines Kupferkations gelöscht wird. Die reversible Reaktion ist dann durch kurze zufällige Intervalle hoher und niedriger Fluoreszenzemission gekennzeichnet. Hohe Fluoreszenzemission entspricht dem freien Liganden, während dunkle Zustände den zugehörigen Kupferkomplex signalisieren. Inzwischen haben wir diese Arbeiten auf kompliziertere chemische Reaktionen ausgeweitet. Die Arbeiten umfassen Versuche mit Redoxsonden zur direkten Beobachtung von Redoxreaktionen sowie katalytischen Reaktionen an immobilisierten Übergangsmetallkomplexen.
Kürzlich ist es uns gelungen, die Kupfersonde im Bereich der hoch auflösenden Mikroskopie einzusetzen. Bis vor wenigen Jahren wurde das von Ernst Abbe erstmals im Jahr 1873 beschriebene Auflösungsvermögen optischer Mikroskope als unüberwindbar angesehen. Inzwischen sind verschiedene Verfahren bekannt, mit denen diese sogenannte Beugungsgrenze umgangen werden kann.
Eines dieser Verfahren basiert auf der mikroskopischen Abbildung der Fluoreszenz einzelner Moleküle. Hierbei nutzt man die Tatsache, dass die Genauigkeit der Positionsbestimmung eines einzelnen Moleküls um ein bis zwei Größenordnungen besser ist als die Grenze der von Abbe definierten optischen Auflösung, die rund 0,2 Mikrometer beträgt. Liegen zwei Farbstoffmoleküle aber zu nah beieinander, überlagern sich ihre Abbildungen derart, dass sie nicht mehr aufgelöst werden können.
Die neuen Methoden umgehen das Problem der Überlagerung, indem sie die Fluoreszenz der Moleküle durch zusätzliche Einstrahlung von Licht geeigneter Wellenlänge aus- beziehungsweise anschalten. Dadurch erreicht man, dass in jedem Bild nur eine kleine, zufällige Anzahl von Farbstoffen fluoresziert. Deren Position kann nun viel genauer bestimmt werden. Durch wiederholtes An- und Ausschalten werden in zufälliger Weise alle Farbstoffe durchgeschaltet und – wie in der Abbildung rechts gezeigt – jeweils einzeln abgebildet.
Wir konnten dieses Verfahren wesentlich vereinfachen, indem wir das lichtgesteuerte Schalten durch eine reversible chemische Reaktion – nämlich die oben beschriebene Komplexierung durch Kupferkationen – ersetzt haben. Bei einer hinreichend hohen Konzentration von Kupferkationen sind fast alle Sonden ausgeschaltet. Damit ist es möglich, die exakte Position der wenigen verbliebenen fluoreszierenden Sonden zu bestimmen. Die reversible Reaktion schaltet in der Folge zufällig andere Sonden an beziehungsweise aus, was wir mit einer CCD-Kamera aufzeichnen. Aus den entstandenen Filmen bestimmen wir die exakten Positionen, die in eine Rekonstruktion eingetragen werden (siehe Abbildung oben). Das rekonstruierte Bild zeigt eine um den Faktor 10 erhöhte Auflösung gegenüber der herkömmlichen fluoreszenzmikroskopischen Aufnahme.
Die beschriebenen Arbeiten deuten auf ein hohes Potenzial von Fluoreszenzsonden hin. Das liegt nicht zuletzt an der Empfindlichkeit der Fluoreszenzdetektion, die zu den empfindlichsten Verfahren gehört, die unter normalen Bedingungen eingesetzt werden können. Darüber hinaus lassen sich spektroskopische Eigenschaften, die sich in der Änderung der Fluoreszenz widerspiegeln, in analoger Art und Weise mit molekularen Eigenschaften verknüpfen. Dies kann die Grundlage für neue Marker und Sonden sein, die über die Mikroskopie hinaus auch Potenzial für die medizinische Diagnostik aufweisen.
Schließlich zeigen die Beispiele, dass die Entwicklung neuer Fluoreszenzsonden nicht nur neue Einblicke in molekulare Prozesse erlaubt, sondern als Nebenprodukt auch die Entwicklung neuer Methoden, wie am Beispiel der hochauflösenden Mikroskopie gezeigt. Dabei haben wir die Erfahrung gemacht, dass gerade die interdisziplinäre Zusammenarbeit zwischen Physikern, Chemikern, Biologen und Medizinern zu immer neuen wissenschaftlichen Perspektiven führt.

| |
|
Foto: Friederike Hentschel
|
Michael Schwering (links) studierte Physik in Bielefeld und promoviert seit dem Jahr 2008 in der Arbeitsgruppe von Dirk-Peter Herten.
Kontakt: michael.schwering@bioquant.uni-heidelberg.de
Anne Seefeld (mitte) studierte Biologie und Chemie an der Universität Heidelberg. Seit Dezember 2010 ist sie in der Arbeitsgruppe von Dirk-Peter Herten als Doktorandin tätig. Ihr Forschungsfeld umfasst die Entwicklung molekularer Sonden für die Einzelmolekül-Spektroskopie und deren Anwendung in biologischen Systemen.
Priv.-Doz. Dr. Dirk-Peter Herten (rechts) forscht seit dem Jahr 2007 im Bereich der Einzelmolekül-Spektroskopie als Leiter einer unabhängigen Nachwuchsgruppe im Exzellenzcluster „Zelluläre Netzwerke“ der Universität Heidelberg. Vorher arbeitete er bei einem Startup-Unternehmen im Bereich der Geräteentwicklung, bevor er 2003 in das Physikalisch-Chemische Institut der Universität Heidelberg wechselte, wo er im Jahr 2008 am Lehrstuhl von Prof. Dr. Jürgen Wolfrum habilitierte.
Kontakt: dirk-peter.herten@urz.uni-heidelberg.de

