Eine Irrfahrt im Raum?
Das Erbmolekül DNS aus der Sicht der Physik
von Dieter W. Heermann
Eine der größten Aufgaben der modernen Physik ist es, eine „Physik des Lebens“ zu entwickeln. Was es zu verstehen gilt, ist eine ebenso schwierige wie grundlegende Frage: Wie entsteht aus unbelebter Materie – also aus Atomen – Leben? Das ist eine gewaltige Herausforderung. Und man kann sich einer Lösung der Frage nur in kleinen Schritten nähern.
| |
|
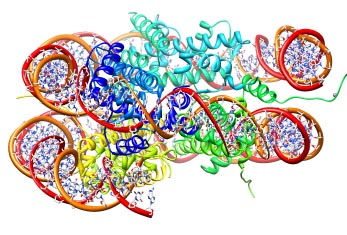
Die erste „Verpackungseinheit“ ist das Nukleosom, ein Komplex aus DNS und Proteinen.
|
Lebende Systeme sind meist durch Unordnung charakterisiert. Zudem entsteht in der belebten Materie Funktionalität nur im Zusammenspiel – die Funktion verschwindet, isoliert man Bausteine voneinander. Aus der Sicht der Physik besteht die Herausforderung also genauer betrachtet darin, ein nicht isoliertes System zu verstehen, in dem Unordnung herrscht.
Die physikalische Struktur des Erbmoleküls DNS (Desoxyribonukleinsäure) wurde durch die berühmten Experimente von Crick und Watson aufgeklärt. Betrachtet man die isolierte DNS, dann handelt es sich bei ihr um eine Aneinanderreihung von Molekülen (Monomere) zu einer Polymerkette. Die Methoden, die in der Polymerphysik entwickelt wurden, sollten also auch auf die DNS anzuwenden sein. So zeigt die isolierte DNS-Kette das Verhalten eines „Self-Avoiding Walk“ – einer Irrfahrt mit Selbstvermeidung. Im lebenden System gesellen sich zur DNS noch andere Moleküle hinzu, beispielsweise Histone, das sind Proteine, welche die DNS „verpacken“. Mit noch weiteren Molekülen geht die DNS Wechselwirkungen ein. Und auch das beeinflusst ihre physikalischen Eigenschaften.
Wir wollen diesem Umstand nachgehen, indem wir die DNS auf verschiedenen Längenskalen untersuchen und nach ihrer physikalischen Ordnungsstruktur fragen. Und wir tun dies in einem bestimmten Zustand im Leben einer Zelle, der sogenannten Interphase des Zellzyklus.
| |
|
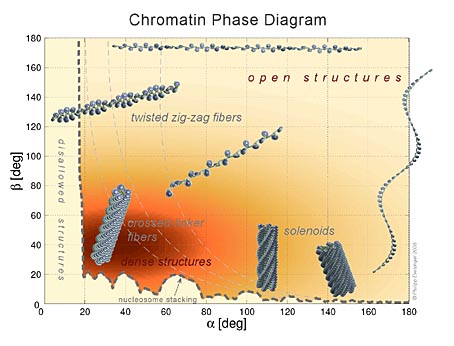
Das Phasendiagramm zeigt alle physikalisch möglichen Chromatin-Strukturen und die Trennlinie zu den nicht möglichen Strukturen. Gezeigt ist der Fall, wenn das Histon H1 mit der Länge Null angesetzt und die Windung der DNS nicht berücksichtigt ist. Wird sie berücksichtigt, werden wesentlich mehr Strukturen möglich.
|
Betrachten wir zunächst die Ordnung auf einer Skala oberhalb der Ordnungsstruktur der DNS. Diese Organisationsstufe ist durch das „Nukleosom“ charakterisiert. Nukleosom heißt: Der DNS-Faden wickelt sich circa 1,65 Mal um ein Histon-Oktamer, man spricht auch von einem „oktameren Nukleosomenkern“. Dieser Kern wird von jeweils zwei Kopien der Histone H2A, H2B, H3 und H4 gebildet.
| |
|
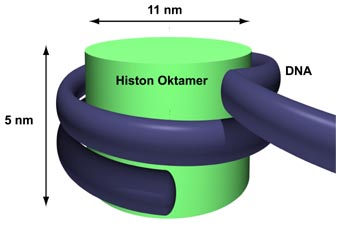
Die nächsthöhere Ordnungsstruktur oberhalb der DNS: Um einen Kern aus Proteinen (grün) – sie werden „Histone“ genannt – wickelt sich das Erbmolekül (blau) und zwar exakt 1,65 Mal.
|
Der oktamere Nukleosomenkern sieht in etwa so aus wie ein Zylinder, der fünf Nanometer hoch ist und einen Durchmesser von elf Nanometern hat. Man darf sich diese Struktur allerdings nicht statisch vorstellen: Die DNS ist nämlich fähig, sich um den oktameren Nukleosomenkern herum zu bewegen. Außerdem können Nukleosomen entlang der DNS fehlen.
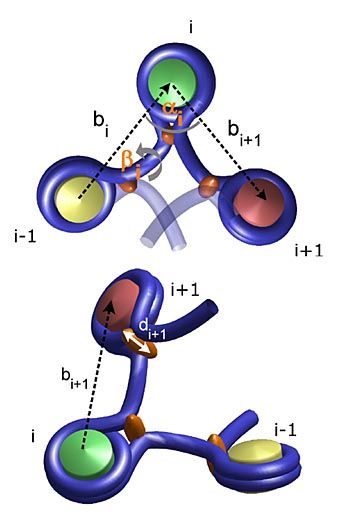
Mit dieser Grundstruktur lässt sich nun eine Kette bauen, das sogenannte Chromatin. Wir nehmen dazu drei Nukleosome und haben dabei Folgendes zu berücksichtigen: den Öffnungswinkel a zwischen drei Nukleosomen (siehe Abbildung auf Seite 17), die Drehung zweier Nukleosome gegeneinander sowie die Länge des DNS-Stücks zwischen zwei Nukleomsomen (der sogenannte Linker). Die Länge des Linker halten wir fest und rechnen diese Größe in die beiden Parameter Öffnungswinkel und Verwindung (Twist) ß ein. Jetzt haben wir noch zu berücksichtigen, dass zwei Nukleosome nicht teilweise oder ganz übereinanderliegen dürfen (ausgeschlossenes Volumen).
| |
|
Mit dem Nukleosom, der fundamentalen Verpackungseinheit der DNS, lässt sich eine Kette bauen: das „Chromatin“. Für das Modell wurden drei Nukleosome gewählt (grün, rot, gelb; braun dar?gestellt ist das Histon H1, das am Aufbau höherer Strukturen beteiligt ist und sie fixiert). Zu berück?sichtigen ist der Öffnungswinkel zwischen den drei Nukleosomen, die Drehung zweier Nukleosome gegeneinander und die Länge des DNS-Fadens (blau) zwischen zwei Nukleosomen. Anschließende Berechnungen ergeben als mögliche Grundmuster der Kette eine Zick-Zack- oder eine spulenartige Solenoid-Struktur.
|
Wenn alle Nukleosome und alle H1-Histone vorhanden sind, ergeben sich mögliche Grundmuster des Chromatins, etwa eine Zick-Zack- oder eine Solenoid-Struktur. Die nächste Frage ist: Welche Struktur findet sich tatsächlich in den Experimenten? Die Situation erweist sich zunächst als recht unübersichtlich: Einige Experimente sprechen für eine Zick-Zack-, andere für eine Solenoid- und wieder andere für überhaupt keine Struktur.
Betrachten wir dies von der theoretischen Seite, stellt sich die Frage nach einem Phasendiagramm, welches die möglichen Strukturen charakterisieren kann. Die Parameter sind der Öffnungswinkel a und die Verwindung ß. Zudem müssen wir noch den Gang der DNS berücksichtigen, also diejenige Strecke, um die sich die DNS bei einer vollen Umdrehung nach oben windet.
Die Abbildung oben zeigt ein solches Phasendiagramm. Es präsentiert alle physikalisch möglichen Strukturen sowie die Trennlinie zu den unmöglichen Strukturen, also denjenigen, wo das Prinzip des ausgeschlossenen Volumens verletzt ist. Bei einer sehr kleinen Ganghöhe der DNS zeigen sich tiefe Mulden. Sie separieren den Öffnungswinkel in diskrete mögliche Werte. Wenn man als Prinzip verlangt, dass die Verwindung ß stets klein ist, dann bedeutet dies eine starke Trennung zwischen mehr Zick-Zack-bestimmten und mehr Solenoid-bestimmten Strukturen.
Interessant wird es, wenn wir den Gang größer werden lassen, hin zu den Werten, die ungefähr der Länge des H1-Histons entsprechen. Bei solchen Werten verschwindet die vorher so prominente Trennung, die Werte für den Öffnungswinkel können nun wesentlich freier variieren. Nun wird eine Mischung aus Zick-Zack- und Solenoid-Struktur möglich.
Was passiert aber, wenn H1 an einigen Stellen eliminiert wird und Histone entfernt werden? Wie die Simulationen deutlich zeigen (siehe die Abbildungen auf Seite 18), wird die Struktur nun wesentlich ungeordneter.
Eine weitere interessante Frage ist, wie die Struktur der gesamten Chromatinkette – also des Chromosoms – aussieht, wenn die Zelle arbeitet. In der sogenannten kondensierten Phase des Zellzyklus, wenn die Chromosomen im Mikroskop sichtbar sind, ist die Struktur relativ klar zu erkennen. Im aktiven Teil des Zellzyklus aber ist jedes Chromosom dekondensiert und mit gewöhnlichen Lichtmikroskopen nicht mehr zu erkennen. Sehr viel Arbeit ist deshalb in die Entwicklung neuer Mikroskope geflossen, um die Auflösung zu erhöhen (siehe auch den Beitrag „Neue Mikroskope überschreiten für unüberwindlich gehaltene Grenzen“ von Christoph Cremer in der Ruperto Carola, Ausgabe 3/2008.)
Eine Möglichkeit, die Struktur eines Chromosoms trotz der immer noch unzulänglichen Auflösung zu erkennen, besteht darin, fluoreszierende Marker am Chromosom zu befestigen. Nehmen wir an, wir befestigen zwei solcher leuchtender Marker im genomischen Abstand „g“ (also der Konturlänge), dann können wir den physikalischen Abstand (also die Distanz der Marker im Raum) messen. Wenn das Chromosom sich wie eine sehr lange Polymerkette verhalten würde, dann würde sich das Abstandsquadrat ungefähr linear mit dem genomischen Abstand verändern. Das Chromosom könnten wir dann als eine Art „Irrfahrt im Raum“ auffassen.
Experimente zeigen, dass der physikalische Abstand ab einem genomischen Abstand von ungefähr 1,5 Mb (Mega-Basenpaaren) nur noch sehr geringfügig zunimmt. Dies widerspricht der Annahme, dass ein Chromosom ein lineares Polymer ist. Im Grunde genommen besteht aber ein Chromosom sehr wohl aus einer linearen Kette, wie wir sie oben beschrieben haben. Wie können wir dennoch das experimentelle Verhalten erklären?
Die zentrale Idee für eine Erklärung ist, dass sich Schleifen bilden. Eine Schleife bedeutet, dass ein oder mehrere Gene miteinander wechselwirken und kolokalisiert sind. Eine Schleife allein reicht jedoch als Erklärung nicht aus. Und es reicht auch nicht aus, Schleifen zu postulieren, die sich wiederholen. Entscheidend ist, dass die Schleifen auf allen Skalen entstehen, damit sich eine Sättigung des physikalischen Abstands als Funktion des genomischen Abstands ergibt.
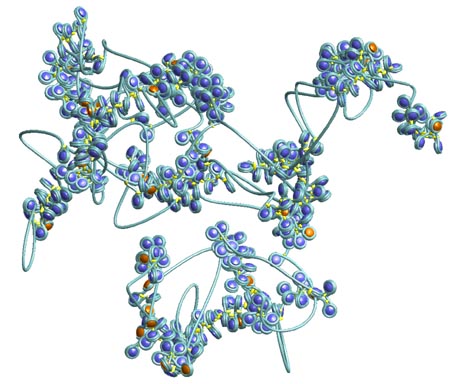
| |
|
Computersimulation einer Chromatinkette. Die rot markierten Nukleosome weisen Defekte im Histon H1 auf. Wenn H1 an einigen Stellen eliminiert wird oder Histone aus dem Nukleosomenkern entfernt werden, entsteht eine wesentlich ungeordnetere Struktur.
|
Halten wir erstens fest: Die Organisationsstruktur des aktiven Chromosoms ist geprägt durch eine komplexe, ineinander verschachtelte Schleifenstruktur. Mehr noch: Die Schleifen müssen sich nicht nur verschachteln, sondern auch stets neu bilden. Nimmt man nun an, die Struktur der Schleifen sei einmal fixiert und verändere sich anschließend nicht mehr, dann ergibt sich eine Diskrepanz zwischen dem theoretischen Modell und den tatsächlich vorliegenden experimentellen Daten. Es muss also stets neue Kolokalisationen zwischen zwei oder mehreren Genen während der Interphase geben, also derjenigen Phase, in der eine Zelle sich nicht teilt, sondern ihre spezifische Funktion ausübt.
Halten wir zweitens fest: Leben entsteht aus geordneten Strukturen – die sich jedoch rasch in ungeordnete Strukturen weiterentwickeln können. Dieses Wechselspiel zu verstehen, bedarf der aktiven Zusammenarbeit von Biologen, Physikern und Mathematikern und des steten Abgleichs von Theorie und Experiment. Eine Physik des Lebens kann nur durch interdisziplinäre Forschung entwickelt werden. Nur, wenn wir Brücken zwischen den Disziplinen bauen, können wir zu neuen Erkenntnissen finden.

| |
|
|
Prof. Dr. Dieter Heermann arbeitet seit dem Jahr 1989 am Institut für Theoretische Physik und am Interdisziplinären Zentrum für Wisschenschaftliches Rechnen (IWR) der Universität Heidelberg. Vorherige Stationen waren Boston, Mainz und Wuppertal. Seit 2007 ist er Mitglied des „Institute for Molecular Biophysics“ des Jackson Laboratory (USA); 2008 wurde er zum Adjunct Professor am Jackson Laboratory berufen.
Kontakt: heermann@tphys.uni-heidelberg.de

