Heidelberger Forscher entschlüsseln wichtigen Teil des zellulären Proteintransportsystems
4. April 2014
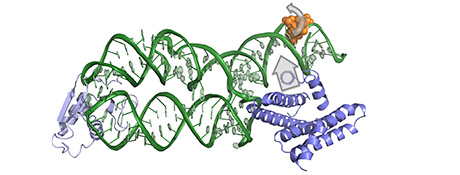
Grafik: K. Wild, Abteilung Strukturbiologie
Mit ihren Forschungen zum zellulären Proteintransport ist es Heidelberger Wissenschaftlern gelungen, ein weiteres wichtiges Element dieses komplexen Transportsystems strukturell und funktionell zu charakterisieren. Im Mittelpunkt steht dabei das sogenannte Signal-Erkennungspartikel, kurz SRP, das als molekularer „Postbote“ für die Sortierung und Membraninsertion von Proteinen sorgt. Das Team um Prof. Dr. Irmgard Sinning vom Biochemie-Zentrum der Universität Heidelberg hat jetzt einen zentralen SRP-Bestandteil entschlüsselt, über den bisher nur wenig bekannt war. Die Ergebnisse dieser Untersuchungen wurden heute in „Science“ veröffentlicht.
Jede Zelle enthält Hunderte von Proteinen, von denen mehr als ein Drittel für den Einbau in Zellmembranen oder den Export aus der Zelle heraus sortiert werden muss. Zum Einsatz kommt dabei ein molekularer „Postbote“ – das SRP. Ohne die SRP-Logistik bricht der zelluläre Verkehr zusammen. Mit Hilfe eines eingebauten Transportsignals werden SRP-Pakete bereits an den Synthesefabriken der Zelle – dies sind die Ribosomen – abgeholt. Von dort aus geht es zum Postausgang, dem Translokations-Kanal. Im menschlichen Organismus ist das SRP ein makromolekularer Komplex, der aus einer Ribonukleinsäure, der SRP-RNA, und sechs daran gebundenen Proteinen besteht. Während mittlerweile vier dieser Proteine im atomaren Detail bekannt sind, haben sich die beiden größten – SRP68 und SRP72 – bisher „hartnäckig einer näheren Untersuchung verweigert“, wie Prof. Sinning sagt.
Der von Irmgard Sinning geleiteten Abteilung Strukturbiologie ist es jetzt gelungen, einen essentiellen Teil des SRP-Systems – die RNA-Bindedomäne von SRP68 – zu charakterisieren. Dabei ging es den Wissenschaftlern um die Frage, wie eben dieses Protein an der SRP-RNA bindet. Wie die Heidelberger Forscher herausgefunden haben, besitzt SRP68 ein sogenanntes argininreiches Motiv (ARM). Dieses sorgt nicht nur für die Bindung, sondern ändert dabei auch signifikant die Struktur der SRP-RNA. Der „starke ARM“ biegt die RNA in ihre funktionsfähige Form. „Ohne diese Änderung könnte das SRP nicht richtig an das Ribosom binden, und der Transport der dort produzierten Proteine zum Translokations-Kanal würde blockiert“, erläutert Prof. Sinning.
Die Analyse früherer elektronenmikroskopischer und biochemischer Daten lässt noch weitere Schlüsse zu: Durch das Verbiegen der RNA werden zwei Basen nach außen gedrückt, die einen direkten Kontakt mit dem Ribosom ausbilden. Bei Erreichen des Translokations-Kanals wird dieser Kontakt wieder aufgelöst, und die beiden Basen stehen für die Regulation des Antriebssystems der Translokation zur Verfügung. „Mit unseren Forschungsarbeiten zum ,starken ARM‘ der Proteintranslokation konnten wir einen der letzten weißen Flecke des SRP-Systems beschreiben“, betont Dr. Klemens Wild aus der Abteilung von Prof. Sinning.
Originalpublikation:
J.T. Grotwinkel, K. Wild, B. Segnitz and I. Sinning: SRP RNA Remodeling by SRP68 Explains Its Role in Protein Translocation, Science (4 April 2014), Vol. 344 no. 6179 pp. 101-104, doi: 10.1126/science.1249094

