Wie Schalter in Bakterien funktionieren
12. Oktober 2017
Forscher aus Karlsruhe, Heidelberg und Berlin analysieren Struktur und Dynamik von Riboschaltern
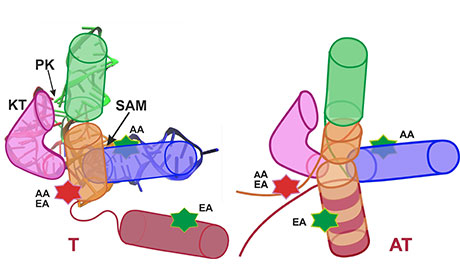
Abbildung: Institut für Angewandte Physik/KIT
Viele Bakterien besitzen Riboschalter als molekulare Kontrollelemente, über die sie Gene an- und abschalten können. Wie diese „riboswitches“ funktionieren, haben Forscher des Karlsruher Instituts für Technologie, der Universität Heidelberg und der Freien Universität Berlin nun mithilfe der lichtoptischen Mikroskopie an Einzelmolekülen grundlegend untersucht. Der Einsatz dieser Schalter könnte beispielsweise neue Möglichkeiten bei der Entwicklung von Antibiotika eröffnen, so der Heidelberger Wissenschaftler Prof. Dr. Andres Jäschke. Die Forschungsergebnisse wurden in der Zeitschrift „Nature Chemical Biology“ veröffentlicht.
Riboschalter liegen auf der Boten-Ribonukleinsäure, die genetische Information zum Ort der Herstellung von Proteinen transportiert. Ein solcher Schalter besteht aus einem Sensor, der die Konzentration eines kleinen Stoffwechselmoleküls misst, und einem Effektor, der die Genexpression und damit die Herstellung eines Proteins steuert. Im Mittelpunkt der aktuellen Untersuchungen stand der SAM-I-Riboschalter. SAM steht dabei für S-Adenosyl-L-Methionin. Die Anbindung des SAM-Moleküls bewirkt bei diesem Schalter, dass sich die räumliche Anordnung der Atome – die Konformation – verändert. Sie wechselt in An- oder Abwesenheit von SAM zwischen der Antiterminator-Struktur (Schalter ein) und der Terminator-Struktur (Schalter aus), abgekürzt AT und T. Dadurch werden Gene an- und abgeschaltet und letztlich die Proteinbiosynthese gesteuert.
Um diese Vorgänge zu entschlüsseln, haben die Biochemiker in Heidelberg die SAM-I-Riboschalter synthetisiert und an verschiedenen Stellen gezielt mit unterschiedlichen Fluoreszenzfarbstoffen in grün und rot markiert. Von den Karlsruher Biophysikern um Prof. Dr. Gerd Ulrich Nienhaus wurden die Schalter mit besonders empfindlichen Lichtmikroskopen untersucht. Mithilfe der lichtoptischen Einzelmolekülexperimenten gelang es, die Dynamik der Konformation direkt zu erfassen. Dazu wurde der grüne Farbstoff zur Aussendung von Licht angeregt. Befand sich in der Nähe der rote Farbstoff, konnte dieser die Anregungsenergie übernehmen und selbst Licht aussenden. Die Wahrscheinlichkeit eines Energietransfers war dabei stark abhängig vom Abstand der Farbstoffe zueinander. Da die Lichtemission jedoch extrem schwach war, bedurfte es aufwendiger Datenanalyseverfahren, die von der Chemikerin Prof. Dr. Bettina Keller in Berlin entwickelt wurden.
Mit ihren Untersuchungen gelang es den Forschern, nicht nur wie erwartet zwei, sondern insgesamt vier Konformationen zu unterscheiden. Überraschend war dabei auch, dass der SAM-I-Riboschalter nicht vollständig zwischen AT- und T-Strukturen hin und her schaltete, sondern ständig zwischen allen Zuständen hin und her fluktuierte. Dabei waren diese Strukturfluktuationen mit angedocktem SAM-Molekül deutlich schneller als ohne SAM-Anbindung. Diese Beschleunigung durch die Bindung von SAM sorgt dafür, dass die T-Struktur (Schalter aus) schnell genug eingestellt werden kann. „Damit wird offenbar sichergestellt, dass das schmale Zeitfenster, in dem die Abschaltung der Genexpression überhaupt möglich ist, effektiv genutzt werden kann“, erläutert Prof. Jäschke, der auf Heidelberger Seite die Forschungsarbeiten leitete. „Das zeigt, dass die Dynamik des SAM-I-Riboschalters eine entscheidende Rolle für seine Funktion besitzt“, so der Wissenschaftler, der Leiter der Forschungsgruppe Bioorganische Chemie und Chemische Biologie am Institut für Pharmazie und Molekulare Biotechnologie ist.
Da Riboschalter in vielen bakteriellen Krankheitserregern vorkommen, stellen sie bei der Entwicklung neuer Antibiotika wichtige Angriffsziele dar. Weitere Anwendungsmöglichkeitem eröffnen sie in der synthetischen Biologie. So könnten Bakterien mit Riboschaltern gentechnisch so modifiziert werden, dass sie niedermolekulare Umweltgifte wie Herbizide aufspüren und abbauen. Die Forschungsarbeiten wurden von der Heidelberg Karlsruhe Research Partnership (HEiKA) gefördert. HEiKA wird von der Universität Heidelberg und dem Karlsruher Institut für Technologie getragen.

