Vorstoß in den Nanokosmos
Neue Mikroskope überschreiten für unüberwindlich gehaltene Grenzen
von Christoph CremerDas Lichtmikroskop hat einst das Tor in eine neue Welt eröffnet. Doch alle Geheimnisse des Mikrokosmos enthüllen konnte es nicht: Strukturen, die kleiner als 200 Nanometer sind, kann Licht nicht sichtbar machen. Neue Methoden erlauben es heute, die bislang für absolut gehaltene Grenze der räumlichen Auflösung zu überwinden und tief, bis in die Nanowelt der Zellen, vorzustoßen.

| |
|
Der Physiker Ernst Abbe begründete die wissenschaftliche Mikroskopie. Der Ausschnitt oben rechts zeigt die berühmte Formel für die Grenze der optischen Auflösung
Foto: S. Hell
|
Der Ursprung des Universums, die Struktur der Materie und die Natur des Lebens sind Grundthemen der Physik – heute wie vor 2500 Jahren. Während die beiden ersten Themen bereits in Astro-, Hochenergie-, Atom-, Molekül- und Festkörperphysik eine weitläufige Heimat gefunden haben, erlauben es die Fortschritte der Molekular- und Zellbiologie heute, sich dem dritten Hauptproblem der Naturerkenntnis mit größerer Aussicht als jemals zuvor zuzuwenden: der Physik des Lebens.
Wie hängen die unvorstellbar komplexen biologischen Strukturen mit den fundamentalen Gesetzen zusammen? Ein wesentlicher Schritt zur Lösung dieses Welträtsels war eine Entdeckung, die der deutsche Botaniker Matthias Schleiden im Jahr 1838 mithilfe der Lichtmikroskopie machte: Jeder Organismus ist „kein Einzelnes, sondern eine Versammlung von lebendigen selbständigen Wesen, die der Idee, der Anlage nach gleich sind“ – so wie Goethe es bereits im Jahr 1807 vermutet und formuliert hatte.
Auch der menschliche Körper ist aus unzähligen solcher „Einzelwesen“ aufgebaut: den Zellen. Die im Innern und zwischen den Zellen ablaufenden Vorgänge entscheiden über Gesundheit und Krankheit, Jugend, Altern und Tod. Zellen stehen daher seit vielen Jahrzehnten im Zentrum biomedizinischer und biophysikalischer Forschung.
Wie hängen die unvorstellbar komplexen biologischen Strukturen mit den fundamentalen Gesetzen zusammen? Ein wesentlicher Schritt zur Lösung dieses Welträtsels war eine Entdeckung, die der deutsche Botaniker Matthias Schleiden im Jahr 1838 mithilfe der Lichtmikroskopie machte: Jeder Organismus ist „kein Einzelnes, sondern eine Versammlung von lebendigen selbständigen Wesen, die der Idee, der Anlage nach gleich sind“ – so wie Goethe es bereits im Jahr 1807 vermutet und formuliert hatte.
Auch der menschliche Körper ist aus unzähligen solcher „Einzelwesen“ aufgebaut: den Zellen. Die im Innern und zwischen den Zellen ablaufenden Vorgänge entscheiden über Gesundheit und Krankheit, Jugend, Altern und Tod. Zellen stehen daher seit vielen Jahrzehnten im Zentrum biomedizinischer und biophysikalischer Forschung.

| |
|
George Seurat malte die „Brücke von Courbevoie“ um das Jahr 1888. So ähnlich wie ein Maler sein Landschaftsbild konstruiert, kann man sich die Abbildung einer mikroskopisch kleinen Struktur vorstellen: Die einzelnen Farbpunkte sind hier die Orte einzelner Moleküle.
|
Typischerweise haben Zellen einen Durchmesser von 0,01 bis 0,02
Millimeter. Sie besitzen an ihrer Oberfläche und in ihrem Inneren
zahlreiche noch viel kleinere „Nano“-Strukturen, deren Eigenschaften
für die Steuerung des Lebens entscheidend sind. Für die Erforschung
solcher Nanostrukturen – sie werden auch „Biomolekulare Maschinen“
genannt – wurden zahlreiche physikalische Verfahren entwickelt, etwa
die Elektronenmikroskopie und die Röntgenkristallographie, oder es
erfolgte die Analyse mithilfe energiereicher Synchrotronstrahlung.
Lichtmikroskopische Methoden waren für die moderne Zellbiologie bis vor kurzem nur von geringerer Bedeutung. Dies lag an der auf wenige hundert Nanometer begrenzten optischen Auflösung (1 Nanometer=1nm=1/1000 Mikrometer=1 Millionstel Millimeter): Diese Grenze galt seit den Arbeiten des deutschen Physikers Ernst Abbe (1873) aufgrund der Wellennatur des Lichts als nicht überwindbar. Viele der entscheidenden zellulären Nanostrukturen haben aber eine sehr viel geringere Größe als diese „konventionelle“ Auflösung von circa 200 Nanometer. Sie konnten daher mithilfe des Lichts nicht analysiert werden, infolgedessen schien es unmöglich, lichtmikroskopisch weitere Struktureinzelheiten zu entdecken.
In den letzten Jahren aber ist es gelungen, Methoden zu entwickeln, die gerade dies versuchen: die bislang für absolut gehaltene Grenze der lichtmikroskopischen Auflösung zu überwinden.
Dem Heidelberger Physiker Stefan Hell, heute Direktor am Max-Planck-Institut für Biophysikalische Chemie in Göttingen, ist beispielsweise ein Mikroskopieverfahren zu verdanken, das eine lichtoptische Auflösung von einzelnen, einander benachbarten Makro- molekülen desselben Typs in der Zelle ermöglicht. Die von Stefan Hell entwickelte „STED“-Mikroskopie beruht auf folgender physikalischer Grundidee: Das Objekt wird mithilfe von zwei in raffinierter Weise fokussierter und kombinierter Laserstrahlen Punkt für Punkt abgetastet; die spezifische Wechselwirkung der beiden Laserstrahlen mit dem Objekt wird sodann ausgenutzt, um einzelne Moleküle zu lokalisieren und sichtbar zu machen.
Ein weiteres, der STED-Mikroskopie komplementäres Verfahren steht Forschern ebenfalls seit einigen Jahren zur Verfügung: Es ist die „Spektrale Präzisionsdistanzmikroskopie“ (SPDM) oder „Spektrale Lokalisationsmikroskopie“. Sie wurde von uns Mitte der 1990er Jahre erstmals für „fernfeldfluoreszenzmikroskopische“ Anwendungen konzipiert und seitdem mit zahlreichen Mitarbeitern und Kollegen realisiert. Auch diese Methode ermöglicht es, zelluläre Nanostrukturen zu analysieren und wesentliche Fragen der molekularen Biophysik und Zellbiologie zu lösen.
Ein Beispiel: Es gibt Hinweise darauf, dass die räumliche Gestalt von Genen für ihre Aktivität oder Inaktivität außerordentlich bedeutsam ist und dass die Gen-Gestalt von bestimmten Substanzen beeinflusst werden kann. Mit lichtoptischen „Nanoskopie“-Methoden sollte es möglich sein, die Änderung der Gestalt von Genen im Zellkern zu erforschen. Dies würde zum einen dazu beitragen, einen fundamentalen Lebensprozess – die Genregulation – besser kennen zu lernen; zum anderen wäre besser zu verstehen, wie man die Aktivität von Genen beeinflussen kann, beispielsweise von gefährlichen „Krebsgenen“, die man möglicherweise mit bestimmten Wirkstoffen abschalten könnte. Oder es könnten gewünschte Gene angeschaltet werden, um beispielsweise Hautzellen in „pluripotente Stammzellen“ zurückzuverwandeln, die genutzt werden könnten, um kranke Gewebe zu erneuern.
Lichtmikroskopische Methoden waren für die moderne Zellbiologie bis vor kurzem nur von geringerer Bedeutung. Dies lag an der auf wenige hundert Nanometer begrenzten optischen Auflösung (1 Nanometer=1nm=1/1000 Mikrometer=1 Millionstel Millimeter): Diese Grenze galt seit den Arbeiten des deutschen Physikers Ernst Abbe (1873) aufgrund der Wellennatur des Lichts als nicht überwindbar. Viele der entscheidenden zellulären Nanostrukturen haben aber eine sehr viel geringere Größe als diese „konventionelle“ Auflösung von circa 200 Nanometer. Sie konnten daher mithilfe des Lichts nicht analysiert werden, infolgedessen schien es unmöglich, lichtmikroskopisch weitere Struktureinzelheiten zu entdecken.
In den letzten Jahren aber ist es gelungen, Methoden zu entwickeln, die gerade dies versuchen: die bislang für absolut gehaltene Grenze der lichtmikroskopischen Auflösung zu überwinden.
Dem Heidelberger Physiker Stefan Hell, heute Direktor am Max-Planck-Institut für Biophysikalische Chemie in Göttingen, ist beispielsweise ein Mikroskopieverfahren zu verdanken, das eine lichtoptische Auflösung von einzelnen, einander benachbarten Makro- molekülen desselben Typs in der Zelle ermöglicht. Die von Stefan Hell entwickelte „STED“-Mikroskopie beruht auf folgender physikalischer Grundidee: Das Objekt wird mithilfe von zwei in raffinierter Weise fokussierter und kombinierter Laserstrahlen Punkt für Punkt abgetastet; die spezifische Wechselwirkung der beiden Laserstrahlen mit dem Objekt wird sodann ausgenutzt, um einzelne Moleküle zu lokalisieren und sichtbar zu machen.
Ein weiteres, der STED-Mikroskopie komplementäres Verfahren steht Forschern ebenfalls seit einigen Jahren zur Verfügung: Es ist die „Spektrale Präzisionsdistanzmikroskopie“ (SPDM) oder „Spektrale Lokalisationsmikroskopie“. Sie wurde von uns Mitte der 1990er Jahre erstmals für „fernfeldfluoreszenzmikroskopische“ Anwendungen konzipiert und seitdem mit zahlreichen Mitarbeitern und Kollegen realisiert. Auch diese Methode ermöglicht es, zelluläre Nanostrukturen zu analysieren und wesentliche Fragen der molekularen Biophysik und Zellbiologie zu lösen.
Ein Beispiel: Es gibt Hinweise darauf, dass die räumliche Gestalt von Genen für ihre Aktivität oder Inaktivität außerordentlich bedeutsam ist und dass die Gen-Gestalt von bestimmten Substanzen beeinflusst werden kann. Mit lichtoptischen „Nanoskopie“-Methoden sollte es möglich sein, die Änderung der Gestalt von Genen im Zellkern zu erforschen. Dies würde zum einen dazu beitragen, einen fundamentalen Lebensprozess – die Genregulation – besser kennen zu lernen; zum anderen wäre besser zu verstehen, wie man die Aktivität von Genen beeinflussen kann, beispielsweise von gefährlichen „Krebsgenen“, die man möglicherweise mit bestimmten Wirkstoffen abschalten könnte. Oder es könnten gewünschte Gene angeschaltet werden, um beispielsweise Hautzellen in „pluripotente Stammzellen“ zurückzuverwandeln, die genutzt werden könnten, um kranke Gewebe zu erneuern.

| |
|
Beispiele für optische Lokalisationen aus dem täglichen Leben: Das linke Bild zeigt eine Abendstimmung über der Insel Kos in der Ägäis, der Heimat von Hippokrates, dem Begründer der wissenschaftlichen Medizin. Das rechte Bild zeigt den Leuchtturm auf Mount Desert Island (Maine), Sitz des Jackson Laboratory, Partner der Universität Heidelberg im „Global Network“ (Ölgemälde von Letizia Mancino).
|
Die Methode, auf der die „spektrale Präzisionsdistanzmikroskopie“ beruht, ist jedem aufmerksamen Menschen bekannt: Stellen Sie sich vor, Sie weilen im Urlaub an einem schönen Sommerabend am Ufer der Ägäis und schauen zu einer griechischen Insel hinüber. In der Dämmerung sehen Sie nur undeutlich verschwommene Umrisse; Sie können jedoch einige ferne Positionen auf der Insel wahrnehmen, sobald sie aufzuleuchten beginnen. Sie können sogar Objekte, die sehr nahe beieinanderliegen, voneinander unterscheiden, wenn die Lichter, die sie ausstrahlen, verschiedene Spektralfarben (grün, rot, blau etc.) haben. Sie können dann sehen, ob es sich um Wohnhäuser oder um Kaufhäuser mit ihren Werbesignalen handelt. Wenn aber alle Lichtquellen auf der Insel die gleiche Spektralfarbe hätten – gibt es dann immer noch Möglichkeiten der Unterscheidung?
Ja, die gibt es, und zwar durch das Blinken des Lichtes. Nehmen wir an, Sie sehen, wie ein erstes Licht auf der Insel aufleuchtet und nach einer gewissen Zeit wieder erlischt: Sie können die Lage der Lichtquelle feststellen, Sie können sogar alle blinkenden Lichtquellen lokalisieren – vorausgesetzt, Sie haben Zeit, am Ufer der Ägäis die Nacht zu verbringen. Wenn Sie sich zudem noch den Ort jedes aufleuchtenden und wieder erlöschenden Lichtes merken, können Sie einen Lageplan einzelner Häuser, Geschäfte und Straßenlampen herstellen – obwohl Sie die Insel selbst in tiefer Dunkelheit gar nicht mehr sehen.
Für das Erstellen eines solchen Lageplanes ist es nicht wichtig, in welcher Reihenfolge die Lichter an- und ausgehen: Ob dies in einer festgelegten oder in einer zufälligen Folge geschieht, ändert nichts an der räumlichen Gesamtinformation. Wichtig ist nur, dass das Objekt in einer bestimmten, von der optischen Auflösung vorgegebenen Zone aufleuchtet und dass zu einem bestimmten Zeitpunkt nur ein einziges Objekt leuchtet – wie bei einem Leuchtturm. Diese Grundideen galt es, in die Nanowelt der Zellen zu übersetzen.
| |
|
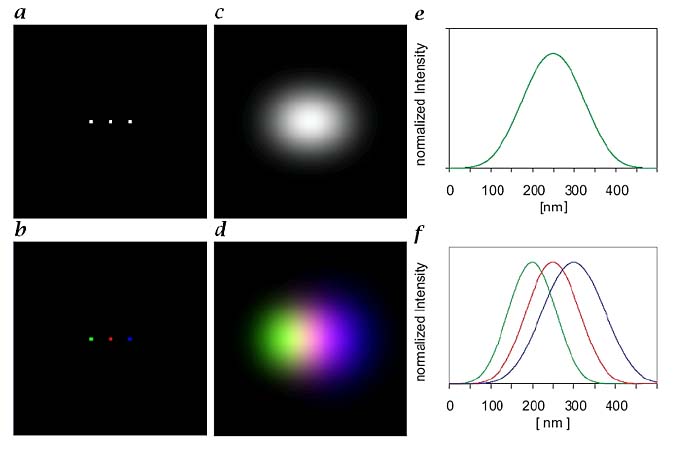
Das Prinzip der spektralen Präzisionsmikroskopie beruht auf einer Entdeckung des britischen Physikers Lord Rayleigh Ende des 19. Jahrhunderts: Er stellte fest, dass das Zentrum der Beugungsscheibe eines punktförmigen Objekts beziehungsweise das Maximum der Helligkeitskurve genau dem Ort des Objekts entspricht.
|
Die Abbildung oben zeigt das Prinzip der spektralen Präzisionsdistanzmikroskopie: Stellen Sie sich drei punktförmig und einfarbig leuchtende Biomoleküle vor, die voneinander einen Abstand von nur 50 Nanometer haben – viermal kleiner als die konventionelle Grenze der optischen Auflösung von circa 200 Nanometer. Von jedem dieser Moleküle wird mithilfe der Mikroskop-Optik und aufgrund der Wellennatur des Lichts ein „Beugungsscheibchen“ erzeugt; diese überlagern sich. Ein Querschnitt durch die Mitte dieses „Beugungsbildes“ ergibt eine glockenförmige Helligkeitskurve, die fast genauso aussieht als ob nur ein einziges Molekül vorhanden wäre. Es ist also nicht möglich festzustellen, wo die drei Moleküle im Bildfeld lokalisiert sind. Wenn aber jedes der eng benachbarten Moleküle eine andere spektrale Signatur hat, wird es möglich, den Ort der einzelnen Moleküle zu bestimmen.
Unter einer spektralen Signatur verstehen wir jede Eigenschaft des von einem punktförmigen Objekt/Molekül ausgesandten Lichts, die es erlaubt, den Ort des betreffenden Objekts/Moleküls mit optischen Mitteln festzustellen und von anderen Molekülen in der Nachbarschaft zu unterscheiden. Dies geschieht zum Beispiel mithilfe verschiedener Spektralfarben.
In der Abbildung oben (b, d) wurde angenommen, dass jedes der zu trennenden eng benachbarten Moleküle eine andere Farbe trägt, etwa grün, rot und blau. Trennt man bei der Aufnahme mittels geeigneter optischer Maßnahmen das grüne, das rote und das blaue Licht voneinander, erhält man drei gegeneinander leicht versetzte Beugungsscheibchen: ein grünes, ein rotes, und ein blaues. Die Mitte des roten Beugungsscheibchens gibt den Ort des rot leuchtenden Moleküls an, die Mitte des blauen Beugungsscheibchens den Ort des blau leuchtenden Moleküls und so fort.
Im Prinzip wurde diese Entdeckung bereits Ende des 19. Jahrhunderts von dem berühmten britischen Physiker Lord Rayleigh gemacht: Er stellte fest, dass das Zentrum des Beugungsscheibchens eines punktförmigen Objekts beziehungsweise das zugehörige Maximum der Helligkeitskurve genau dem Ort des Objekts entspricht. Darauf beruht die Möglichkeit, die Position auch von weit entfernten Leuchttürmen genau festzustellen. Beispielsweise gibt das Zentrum des grünen Beugungsscheibchens in der Abbildung oben den Ort des grün leuchtenden Moleküls an. Da man die Beugungsscheibchen aufgrund ihrer Farben genau voneinander unterscheiden kann, ist es belanglos, wie fern oder wie nah andersfarbige Beugungsscheibchen voneinander liegen: Auf diese Weise kann man den Ort von Molekülen lichtmikroskopisch auch dann noch feststellen, wenn sie – wie in der Abbildung auf Seite 7 – nur einen Abstand von 50 Nanometer haben.
Über Unterschiede in Spektralfarben hinaus gibt es noch weitere Möglichkeiten, eng benachbarte Moleküle lichtoptisch voneinander zu unterscheiden. Wie bei den Lichtern der griechischen Insel könnte man auch die Beugungsscheibchen von eng zusammenliegenden, nur in einer einzigen Spektralfarbe leuchtenden Moleküle voneinander trennen, wenn diese Moleküle ihre Leuchtkraft zeitlich verändern würden: Auch dies ist eine spektrale Signatur.
In den letzten beiden Jahren wurden erstmals in derselben Spektralfarbe leuchtende Moleküle eingesetzt (aber mit verschiedener spektraler Signatur aufgrund von „Blink“-Eigenschaften), die durch Licht in der für die spektrale Präzisionsdistanzmikroskopie erforderlichen Weise an- und abgeschaltet werden können. Durch die Kombination vieler tausender Einzelaufnahmen derselben Zelle wurden mithilfe von laseroptischen Präzisionsmessungen „Lokalisationsbilder“ mit wesentlich verbesserter optischer Auflösung gewonnen. Zu den damit befassten Forscherteams gehören Wissenschaftler der Gruppe um Stefan Hell und des Institute for Molecular Biophysics (Jackson Laboratory, University of Maine). Beide sind der Universität Heidelberg in Projekten der Exzellenzinitiative verbunden.
Die Anwendung dieser neuen Nanoskopie-Verfahren schien bis vor kurzem schwierig, weil angenommen wurde, dass nur speziell hergestellte und mit großem Aufwand einsetzbare Moleküle durch Licht in geeigneter Weise an- und abgeschaltet werden können. Unsere Heidelberger Arbeitsgruppe hat jedoch entdeckt, dass dies unter bestimmten photophysikalischen Bedingungen auch für viele „ganz gewöhnliche“ Farbstoffmoleküle realisiert werden kann. Dies erweitert die Anwendbarkeit der SPDM-Methode auf zahlreiche Gebiete der biophysikalischen, zellbiologischen und medizinischen Forschung.
Darüber hinaus wurde in unserer Arbeitsgruppe am Heidelberger Kirchhoff-Institut für Physik mit der SPDM eine lichtoptische Auflösung (2D) zellulärer Nanostrukturen von etwa zehn Nanometer (=0.000 01 mm) realisiert. Dies entspricht etwa 1/50 der eingesetzten Laserwellenlänge oder dem Durchmesser eines einzelnen Proteins oder 1/1000 Durchmesser eines Zellkerns.
| |
|
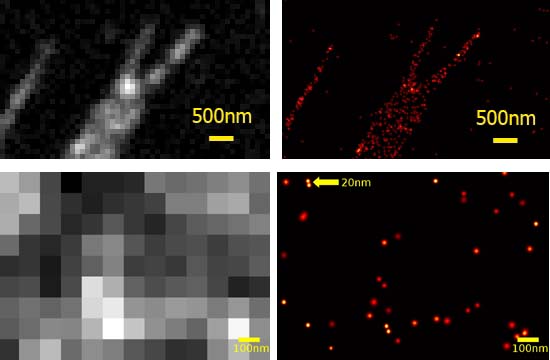
Zum Vergleich: links herkömmliche lichtmikroskopische Bilder – rechts die mit der spektralen Präzisionsmikroskopie erreichbaren Ergebnisse derselben Objekte.
|
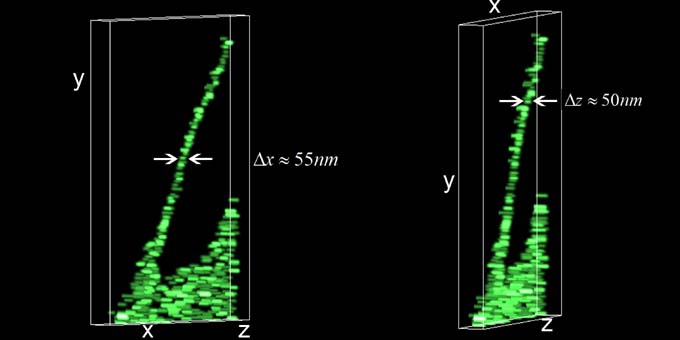
Ein Beispiel wird in der Abbildung oben gezeigt. Zurzeit können wir derartige Bilder mit fast molekularer Auflösung von kompletten Zellen in etwa einer Minute gewinnen. Das hat auch erste SPDM-Aufnahmen von lebenden Zellen erlaubt. Durch die Verbindung mit anderen, ebenfalls in unserer Arbeitsgruppe entwickelten Nanoskopie-Verfahren ist es uns kürzlich sogar gelungen, eine dreidimensionale Auflösung zellulärer Nanostrukturen von circa 40 bis 50 Nanometer zu verwirklichen.
Die Abbildung unten zeigt das dreidimensionale Bild eines kleinen Teils einer menschlichen Krebszelle: Proteine ihrer Membran wurden so markiert, dass ihre Verteilung in den dünnen Verbindungen zu einer benachbarten Krebszelle in allen drei Dimensionen vermessen werden konnte.
Von den zahlreichen Anwendungsmöglichkeiten der spektralen Lokalisationsmikroskopie (SPDM) seien hier exemplarisch folgende genannt:
- die Biophysik der Struktur der im Zellkern gespeicherten genetischen Information;
- die zelluläre Verteilung und Organisation von „biomolekularen Maschinen“;
- die Architektur molekularer Motoren in einzelnen Zellen;
- die funktionelle Genom-Architektur (in molekularer Zellbiologie und medizinischer Forschung);
- die durch ionisierende Strahlung oder andere Umwelteinflüsse verursachte Entstehung krebsrelevanter Genominstabilitäten;
- die Analyse von Membrankomplexen für die Physiologie, beispielsweise zur Erforschung von Herzrhythmusstörungen, für die Immunologie oder die Pharmakologie;
- die Analyse krankheitsrelevanter Proteinaggregate und ihrer Bildung, beispielsweise bei altersbedingten neurobiologischen Degenerationserscheinungen;
- in der Systembiologie die Untersuchung der Nanostruktur molekularer Verbindungsbrücken zwischen den Zellen eines Gewebes oder Tumorgewebes;
- das „Andocken“ von Viren an Zelloberflächen oder die
- Reprogrammierung einzelner Zellen des erwachsenen Organismus zu „pluripotenten“ Stammzellen für die Gewebeerneuerung.
| |
|
Dreidimensionale Nanoskopie einer menschlichen Krebszelle: Mit ihren Ausläufern nimmt sie gerade Kontakt zur Umgebung auf. Die Aufnahmezeit betrug 150 Sekunden.
|
Weltweit ist Heidelberg in der idealen Lage, dass auf seinem Campus die meisten wesentlichen höchstauflösenden Verfahren nicht nur der Lichtmikroskopie/Nanoskopie, sondern auch der Elektronenmikroskopie vorhanden sind. Es ist bemerkenswert, dass viele dieser Geräte hier nicht nur konzipiert, sondern auch entwickelt und gebaut wurden. Dies eröffnet die Möglichkeit einer „Korrelativen Mikroskopie“, bei der zum Beispiel dieselbe zelluläre Nanostruktur zunächst bei molekularer lichtoptischer Auflösung in der lebenden Zelle beobachtet wird, dann schnell fixiert und mit noch höherer Auflösung elektronenmikroskopisch analysiert werden kann. Um solche Voraussetzungen optimal nutzen zu können, ist ein enger Kontakt mit biophysikalischen und biomedizinischen Anwendergruppen essenziell.
Für derartige Projekte gibt es in Heidelberg und Umgebung ein enormes Potenzial. Es reicht vom Max-Planck-Institut für Medizinische Forschung über das European Molecular Biology Laboratory (EMBL), zahlreiche Universitätsinstitute, dem Deutschen Krebsforschungszentrum bis hin zur Medizinischen Fakultät Mannheim der Universität Heidelberg.

| |
|
Foto: Hentschel
|
Prof. Dr. Christoph Cremer
leitet seit 1983 den Bereich „Angewandte Optik und
Informationsverarbeitung“ am Kirchhoff-Institut für Physik sowie seit
2005 zusätzlich den Bereich „Biophysik der Genomstruktur“ am Institut
für Pharmazie und Molekulare Biotechnologie der Universität Heidelberg;
seit 2006 ist er Zweiter Sprecher des Universitätssenats. Zuvor
forschte er an der Universität Freiburg i. Br. und am Lawrence
Livermore National Laboratory/USA. Sein Schwerpunkt ist die Entwicklung
superauflösender Lichtmikroskopiemethoden und deren biophysikalische
Anwendungen.
Kontakt: cremer@kip.uni-heidelberg.de
Telefon: 0 62 21/54 92 71
Kontakt: cremer@kip.uni-heidelberg.de
Telefon: 0 62 21/54 92 71
Seitenbearbeiter:
E-Mail


