T. Beier, C. Pape, N. Rahaman, T. Prange, S. Berg, D.D. Bock, A. Cardona, G.W. Knott, S.M. Plaza, L.K. Scheffer, U. Koethe, A. Kreshuk, and F.A. Hamprecht: Multicut brings automated neurite segmentation closer to human performance, Nature Methods 14, 101-102 (published online 31 January 2017), doi:10.1038/nmeth.4151
Schaltplan des Gehirns
Eine genaue Kenntnis der Verknüpfungen im Gehirn – der Verbindungen zwischen allen Nervenzellen – gilt als Voraussetzung für ein besseres Verständnis dieses komplexesten aller Organe. Wissenschaftler der Universität Heidelberg haben jetzt einen neuen Algorithmus entwickelt, sprich ein neues Rechenverfahren, das mit weitaus größerer Genauigkeit als bisher in der Lage ist, aus mikroskopischen Bildern des Gehirns auf dessen Verknüpfungsstruktur zu schließen.
Von dieser automatischen Auswertung der Bilddaten erwartet Prof. Dr. Fred Hamprecht, Leiter der Arbeitsgruppe „Bildanalyse und Lernen“ am Interdisziplinären Zentrum für Wissenschaftliches Rechnen, wichtige Fortschritte für die Neurowissenschaften: Sie soll zu einem Schaltplan des Gehirns führen. Denn die Funktionsweise des Zentralnervensystems zu verstehen ist eines der großen ungelösten Probleme der Wissenschaft. „Bis heute ist, von einem einfachen Fadenwurm einmal abgesehen, noch nicht einmal der Schaltplan eines kompletten tierischen Gehirns verfügbar, nicht zu reden vom Hirn des Menschen“, erklärt Hamprecht.In den vergangenen Jahren wurden Bildgebungsverfahren entwickelt, die erstmals ausreichend hoch aufgelöste und dreidimensionale Aufnahmen des gesamten Gehirns gestatten. Diese Aufnahmen sind jedoch so groß, dass eine manuelle Analyse Jahrhunderte dauern würde. Gefordert ist daher eine automatische Auswertung mit möglichst geringer Fehlerrate.
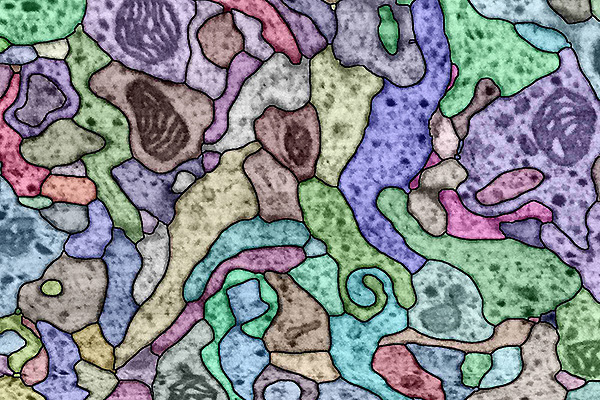
| Die Abbildung zeigt einen winzig kleinen Teil eines Fliegengehirns: Automatische Analyse zerlegt einen dreidimensionalen Bildstapel in einzelne Nervenzellen, die hier mit willkürlichen Farben versehen sind. 20 solcher Bilder nebeneinander entsprechen in etwa dem Durchmesser eines menschlichen Haares. | Abbildung: Thorsten Beier, IWR |
Der neue Heidelberger Algorithmus verwendet nichtlokale Bildinformation: Die Wissenschaftler betrachten Bildregionen, die nicht nebeneinanderliegen, und schätzen, ob sie zur selben Nervenzelle gehören. Wie Wechselwirkungen mit kurzer und langer Reichweite gleichzeitig berücksichtigt werden können, hat Dr. Björn Andres vom Max-Planck-Institut für Informatik in Saarbrücken aufgezeigt. Ziel ist es, eine optimale Lösung zu finden, die beiden Arten von Bildinformationen in bestmöglicher Weise gerecht wird. „Wir erreichen damit erheblich niedrigere Fehlerraten als alle bislang bekannten Methoden“, betont Fred Hamprecht.
Die Herausforderung, mithilfe von Analyse-Verfahren das Verknüpfungsdiagramm eines Gehirns zu erstellen, erläutert Hamprecht am Beispiel der Fliege, die zu erstaunlichen Leistungen fähig ist: Sie findet Futter, Schutz und Geschlechtspartner in einer komplexen und oft feindlichen Umwelt. „Obwohl ihr Gehirn kleiner ist als der Kopf einer Stecknadel, ist das Diagramm ihrer neuronalen Verknüpfungen nach wie vor unbekannt.“ Mit ihrem neuen Algorithmus arbeiten die Heidelberger Wissenschaftler nun zunächst an einem Schaltplan des Fliegenhirns, um sich dann später höheren Tieren zuzuwenden, wie die Mathematikerin Dr. Anna Kreshuk ausführt.
Die Leistungsfähigkeit automatischer Auswertungsverfahren messen Forschergruppen übrigens weltweit in Wettbewerben: Ein vorgegebenes dreidimensionales Bild muss dabei in alle Nervenzellen zerlegt werden, die darauf abgebildet sind. In einem aufwändigen manuellen Verfahren wird zuvor die korrekte – aber geheim gehaltene – Zerlegung ermittelt. Alle eingereichten Ergebnisse werden damit verglichen und das Verfahren mit der niedrigsten Fehlerrate gewinnt. Den Forschern am Interdisziplinären Zentrum für Wissenschaftliches Rechnen ist es jetzt gelungen, im jüngsten Wettbewerb dieser Art, der CREMI Challenge (MICCAI Challenge on Circuit Reconstruction from Electron Microscopy Images), die weitaus höchste Genauigkeit bei der Auswertung zu erzielen.
Die Arbeitsgruppe „Bildanalyse und Lernen“ entwickelt seit 15 Jahren Algorithmen für maschinelle Lernverfahren auf dem Feld „Computer Vision“, die vor allem in den Lebenswissenschaften, aber auch in der Industrie eingesetzt werden. Die jüngsten Forschungsergebnisse, die auf einer engen internationalen Zusammenarbeit beruhen, wurden in der Fachzeitschrift „Nature Methods“ veröffentlicht.
