Eine schützende Kappe für die RNA von Bakterien
3. August 2016
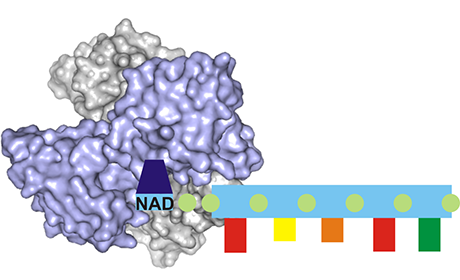
Bild: Katharina Höfer/IPMB
Die Funktionsweise eines sogenannten Decapping-Enzyms in Bakterien haben jetzt erstmals Wissenschaftler der Universität Heidelberg entschlüsselt: Dabei handelt es sich um einen molekularen Helfer, der eine schützende Kappe – oder auch „Cap“ – am Anfang der RNA entfernt. Durch das Decapping wird die Ribonukleinsäure destabilisiert, so dass ihr Abbau in der Zelle eingeleitet werden kann. Während diese Vorgänge für die Boten-RNA in Zellen höherer Organismen gut erforscht sind, wurde dieser Prozess von Prof. Dr. Andres Jäschke und seiner Arbeitsgruppe Bioorganische Chemie nun für bakterielle RNA beschrieben. Bislang ist die Wissenschaft davon ausgegangen, dass Bakterien diese Kappen-Struktur grundsätzlich nicht besitzen. Die Forschungsergebnisse wurden in „Nature Chemical Biology“ veröffentlicht.
Ribonukleinsäuren – in der englischen Abkürzung RNA – sind in Zellen vor allem dafür verantwortlich, dass sie als Boten- oder Gerüstmoleküle dienen, aber auch zentrale biochemische Reaktionen beschleunigen und Stoffwechselprozesse regulieren. In höheren Organismen, den Eukaryonten, trägt die Boten- oder Messenger-RNA (mRNA) an ihrem Anfang üblicherweise eine molekulare Kappe. Diese chemische Modifizierung stabilisiert die Boten-RNA und schützt sie damit vor Abbau und Veränderung. Anders als bei Eukaryonten fehlt diese Kappen-Struktur bei der RNA von Bakterien – so die gängige wissenschaftliche Lehrmeinung. Das Forscherteam unter der Leitung von Prof. Jäschke hat jedoch 2015 entdeckt, dass bei bestimmten bakteriellen Ribonukleinsäuren eine Modifizierung existiert, die der Kappe auf der Boten-RNA bei Eukaryonten strukturell ähnelt.
Bei diesem „Cap“ handelt es sich um das Nicotinamid-Adenin-Dinukleotid (NAD) – ein sogenanntes Coenzym, das im Stoffwechsel von Bedeutung ist. Kommt das NAD hingegen als Kappe der Ribonukleinsäure zum Einsatz, schützt es die RNA gegen Abbau und Modifizierung. Wird die NAD-Kappe entfernt, kann die Ribonukleinsäure dagegen abgebaut werden, um Stoffwechselprozesse einzuleiten. Für den Vorgang des Decapping – die Abspaltung der NAD-Kappe – ist ein Enzym mit dem der Bezeichnung NudC verantwortlich, das von dem Team um Prof. Jäschke als Decapping-Enzym identifiziert worden ist. Die Heidelberger Wissenschaftler vom Institut für Pharmazie und Molekulare Biotechnologie haben nun NudC aus dem Bakterium Escherichia coli mit Hilfe hochaufgelöster Kristallstrukturen analysiert und auf diesem Weg die Funktionsweise dieses Enzyms entschlüsselt.
Die strukturellen Untersuchungen eröffnen ein neues Forschungsgebiet, wie Prof. Jäschke betont, da mögliche Interaktionspartner von NudC ebenso gesucht werden wie auch andere Decapping-Enzyme aus unterschiedlichen Bakterien. Die Wissenschaftler erhoffen sich von den aktuellen Erkenntnissen neue Impulse für die Identifikation unbekannter Kappen-Strukturen sowie deren Funktionsmechanismen in anderen Mikroorganismen.

